给你一棵 n 个节点的有根树,节点编号从 0 到 n - 1 。每个节点的编号表示这个节点的 独一无二的基因值 (也就是说节点 x 的基因值为 x)。两个基因值的 基因差 是两者的 异或和 。给你整数数组 parents ,其中 parents[i] 是节点 i 的父节点。如果节点 x 是树的 根 ,那么 parents[x] == -1 。
给你查询数组 queries ,其中 queries[i] = [nodei, vali] 。对于查询 i ,请你找到 vali 和 pi 的 最大基因差 ,其中 pi 是节点 nodei 到根之间的任意节点(包含 nodei 和根节点)。更正式的,你想要最大化 vali XOR pi 。
请你返回数组 ans ,其中 ans[i] 是第 i 个查询的答案。
示例 1:
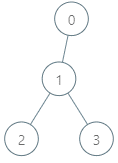
输入:parents = [-1,0,1,1], queries = [[0,2],[3,2],[2,5]] 输出:[2,3,7] 解释:查询数组处理如下: - [0,2]:最大基因差的对应节点为 0 ,基因差为 2 XOR 0 = 2 。 - [3,2]:最大基因差的对应节点为 1 ,基因差为 2 XOR 1 = 3 。 - [2,5]:最大基因差的对应节点为 2 ,基因差为 5 XOR 2 = 7 。
示例 2:
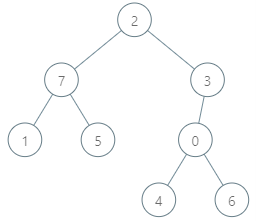
输入:parents = [3,7,-1,2,0,7,0,2], queries = [[4,6],[1,15],[0,5]] 输出:[6,14,7] 解释:查询数组处理如下: - [4,6]:最大基因差的对应节点为 0 ,基因差为 6 XOR 0 = 6 。 - [1,15]:最大基因差的对应节点为 1 ,基因差为 15 XOR 1 = 14 。 - [0,5]:最大基因差的对应节点为 2 ,基因差为 5 XOR 2 = 7 。
提示:
2 <= parents.length <= 105- 对于每个 不是 根节点的
i,有0 <= parents[i] <= parents.length - 1。 parents[root] == -11 <= queries.length <= 3 * 1040 <= nodei <= parents.length - 10 <= vali <= 2 * 105