-
Notifications
You must be signed in to change notification settings - Fork 6
/
R 강의3.Rmd
610 lines (434 loc) · 13.6 KB
/
R 강의3.Rmd
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
88
89
90
91
92
93
94
95
96
97
98
99
100
101
102
103
104
105
106
107
108
109
110
111
112
113
114
115
116
117
118
119
120
121
122
123
124
125
126
127
128
129
130
131
132
133
134
135
136
137
138
139
140
141
142
143
144
145
146
147
148
149
150
151
152
153
154
155
156
157
158
159
160
161
162
163
164
165
166
167
168
169
170
171
172
173
174
175
176
177
178
179
180
181
182
183
184
185
186
187
188
189
190
191
192
193
194
195
196
197
198
199
200
201
202
203
204
205
206
207
208
209
210
211
212
213
214
215
216
217
218
219
220
221
222
223
224
225
226
227
228
229
230
231
232
233
234
235
236
237
238
239
240
241
242
243
244
245
246
247
248
249
250
251
252
253
254
255
256
257
258
259
260
261
262
263
264
265
266
267
268
269
270
271
272
273
274
275
276
277
278
279
280
281
282
283
284
285
286
287
288
289
290
291
292
293
294
295
296
297
298
299
300
301
302
303
304
305
306
307
308
309
310
311
312
313
314
315
316
317
318
319
320
321
322
323
324
325
326
327
328
329
330
331
332
333
334
335
336
337
338
339
340
341
342
343
344
345
346
347
348
349
350
351
352
353
354
355
356
357
358
359
360
361
362
363
364
365
366
367
368
369
370
371
372
373
374
375
376
377
378
379
380
381
382
383
384
385
386
387
388
389
390
391
392
393
394
395
396
397
398
399
400
401
402
403
404
405
406
407
408
409
410
411
412
413
414
415
416
417
418
419
420
421
422
423
424
425
426
427
428
429
430
431
432
433
434
435
436
437
438
439
440
441
442
443
444
445
446
447
448
449
450
451
452
453
454
455
456
457
458
459
460
461
462
463
464
465
466
467
468
469
470
471
472
473
474
475
476
477
478
479
480
481
482
483
484
485
486
487
488
489
490
491
492
493
494
495
496
497
498
499
500
501
502
503
504
505
506
507
508
509
510
511
512
513
514
515
516
517
518
519
520
521
522
523
524
525
526
527
528
529
530
531
532
533
534
535
536
537
538
539
540
541
542
543
544
545
546
547
548
549
550
551
552
553
554
555
556
557
558
559
560
561
562
563
564
565
566
567
568
569
570
571
572
573
574
575
576
577
578
579
580
581
582
583
584
585
586
587
588
589
590
591
592
593
594
595
596
597
598
599
600
601
602
603
604
605
606
607
608
609
610
---
title: "R강의3-기본적인 통계"
author: "문건웅"
output: html_document
---
```{r setup, include=FALSE}
knitr::opts_chunk$set(echo = TRUE,comment = NA)
```
# 지난 주 숙제
(문제)
학생 10명이 있다.영어 시험 결과를 eng, 수학 시험 결과를 math라고 하자.
영어시험은 2번과 9번 학생이 결시하였고 9번 학생이 수학시험을 결시하였다. R에서 결측치는 NA이다.
```{r}
번호=1:10
영어=c(70,NA,80,85,90,90,60,55,NA,90)
수학=c(80,73,84,90,95,85,60,50,NA,100)
data=data.frame(번호,영어,수학)
data
```
위의 성적 data를 사용하여 누락된 값을 각 과목의 평균치로 채워 넣으세요.
(힌트) is.na()와 apply()함수를 사용하세요.
(답)
1. x라는 벡터의 결측치를 평균값으로 바꾸어 본다.
```{r}
x=c(70,NA,80,85,90,90,60,55,NA,90)
x[is.na(x)]=mean(x,na.rm=TRUE)
x
```
2. 이것을 replaceNA라는 함수로 만든다.
```{r}
replaceNA=function(x){
x[is.na(x)]=mean(x,na.rm=TRUE)
x
}
```
3.이것을 apply함수를 이용하여 열 단위로 적용한다.
```{r}
apply(data,2,replaceNA)
```
# 오늘 다룰 내용
* 패키지 설치,이용
* 인터넷에 있는 자료 읽기
* 엑셀(*.xls), csv(*csv) , SPSS(*.sav)화일 읽기
* 한글데이타 읽기
* 평균 검정 ; t-test, ANOVA, two-way ANOVA, ANCOVA
* mytable()
# CRAN의 이용
R-project 홈페이지
[https://www.r-project.org](https://www.r-project.org)
# CRAN에 있는 패키지의 설치
```{r,eval=FALSE}
install.packages("moonBook")
install.packages(c("ztable","webr"))
```
# Github에 있는 패키지의 설치
```{r,eval=FALSE}
install.packages("devtools")
devtools::install_github("cardiomoon/moonBook")
devtools::install_github("cardiomoon/ztable")
devtools::install_github("cardiomoon/webr")
```
# 패키지 불러오기
```{r}
require(moonBook) # library(moonBook)
require(ztable)
require(webr)
```
# 인터넷에 있는 자료 읽기
[Baltimore Camera Data](https://data.baltimorecity.gov/Transportation/Baltimore-Fixed-Speed-Cameras/dz54-2aru)

```{r,eval=FALSE}
if (!file.exists("data")) {
dir.create("data")
}
fileUrl <- "https://data.baltimorecity.gov/api/views/dz54-2aru/rows.csv?accessType=DOWNLOAD"
download.file(fileUrl, destfile = "./data/cameras.csv", method = "curl")
dateDownloaded <- date()
```
# 자료의 읽기 - read.table()
* This is the main function for reading data into R
* Flexible and robust but requires more parameters
* Reads the data into RAM - big data can cause problems
* Important parameters file, header, sep, row.names, nrows
* Related: read.csv(), read.csv2()
* read.csv sets sep="," and header=TRUE
# Example: Baltimore camera data
```{r}
cameraData <- read.table("./data/cameras.csv", sep = ",", header = TRUE)
head(cameraData)
```
# Example: Baltimore camera data
read.csv sets sep="," and header=TRUE
```{r}
cameraData <- read.csv("./data/cameras.csv")
head(cameraData)
```
# Some more important parameters
* quote - you can tell R whether there are any quoted values quote="" means no quotes.
* na.strings - set the character that represents a missing value.
* nrows - how many rows to read of the file (e.g. nrows=10 reads 10 lines).
* skip - number of lines to skip before starting to read
In my experience, the biggest trouble with reading flat files are quotation marks ` or " placed in data values, setting quote="" often resolves these.
# 한글 데이타를 읽을 때 주의점
한글 encoding 은 둘 중의 하나로 되어있다.
* EUC-KR
* UTF-8
한글데이타의 예
```{r}
names=c("김규민","박은혜","서지수")
sex=c("M","F","F")
data=data.frame(이름=names,성별=sex)
data
write.csv(data,"test.csv")
test=read.csv("test.csv",fileEncoding="euc-kr")
test
test=read.csv("test.csv",fileEncoding="utf-8")
test
```
# SPSS 자료 읽기
```{r ,eval=FALSE}
library(foreign)
read.spss("file_name")
```
# 자료를 새로 만들때
## comma seperated file ("*.csv") 로 만들자 !!
# 지난주 복습
```{r}
head(acs,10)
str(acs)
summary(acs)
```
# 데이타 요약; 테이블만들기, 카이제곱,피셔검정
```{r}
table(acs$sex,acs$DM)
result=table(acs$sex,acs$DM)
result
chisq.test(result)
chisq.test(result,correct=FALSE)
fisher.test(result)
#xtabs(도수~가로+세로)
result1=xtabs(~sex+DM,data=acs)
result1
addmargins(result1)
chisq.test(result1)
fisher.test(result1)
```
# p 값이란 무엇인가?
[https://ko.wikipedia.org/wiki/유의_확률](https://ko.wikipedia.org/wiki/유의_확률)
# chisq.test의 해석
[http://r-graph.com:3838/ttest](http://r-graph.com:3838/ttest)
# Yate's correction ?
[https://en.wikipedia.org/wiki/Yates%27s_correction_for_continuity](https://en.wikipedia.org/wiki/Yates%27s_correction_for_continuity)
# 평균의 비교
1) One-sample t-test : 알려져 있는 평균과 비교
2) paired t-test vs Wilcoxon Signed-rank test
3) 두 군 간의 평균 비교 : t.test vs Wilcoxon Rank-sum test
4) 세 군 이상 의 평균비교 : one-way ANOVA vs Kruskall-Wallis test
# One-sample t-test : 알려져 있는 평균과 비교
2006년 조사에 의하면 한국인의 1인 1일 평균 알콜섭취량이 8.1g 이다.
2008년 대통령 선거로 알코올 섭취량이 달라졌는지 조사하였다.
```{r}
x=c(15.5,11.21,12.67,8.87,12.15,9.88,2.06,14.50,0,4.97)
mean(x)
sd(x)
```
검정 순서
1) 자료가 정규 분포를 하는지 검정 : shapiro.test()
2) 정규분포 하는 경우 : t.test()
3) 정규분포 하지 않는 경우 : Wilcoxon Signed-rank test
```{r}
shapiro.test(x)
t.test(x,mu=8.1)
```
단측검정의 경우 : 귀무가설 mu > 8.1, 유의수준 99%
```{r}
t.test(x,mu=8.1,conf.level=0.99,alter="greater")
```
# Paired t-test vs Wilcoxon Signed-rank test
쌍을 이룬 두 변수의 차이를 보는 검정
t-test의 가정 : 자료가 정규분포를 한다
```
검정 순서
1) 자료가 정규 분포를 하는지 검정(shapiro.test())
2) 정규분포 하는 경우 : t.test
3) 정규분포 하지 않는 경우 : Wilcoxon Signed-rank test
```
```{r}
data(sleep)
sleep
str(sleep)
```
## 데이터 sleep 도움말
```{r}
?sleep
```
A data frame with 20 observations on 3 variables.
[, 1] extra numeric increase in hours of sleep
[, 2] group factor drug given
[, 3] ID factor patient ID
## 정규성 검정
```{r}
with(sleep,
shapiro.test(extra[group == 2] - extra[group == 1]))
```
정규분포를 따르지 않으므로 wilcoxon test실시
```{r}
with(sleep,
wilcox.test(extra[group == 2] - extra[group == 1]))
```
비모수방법은 자료의 순서를 이용하는데 동일한 값이 있어 순서를 정하는 데
문제가 있어 warning생김 : exact=FALSE 옵션을 추가한다
```{r}
with(sleep,
wilcox.test(extra[group == 2] - extra[group == 1],exact=FALSE))
```
정규분포를 따르는 경우 Student's paired t-test 실시
```{r}
with(sleep,
t.test(extra[group == 1],
extra[group == 2], paired = TRUE))
```
약을 1에서 2로 바꾸었을때 수면시간이 늘어나는가? The sleep *prolongations*
```{r}
sleep1 <- with(sleep, extra[group == 2] - extra[group == 1])
summary(sleep1)
stripchart(sleep1, method = "stack", xlab = "hours",
main = "Sleep prolongation (n = 10)")
boxplot(sleep1, horizontal = TRUE, add = TRUE,
at = .6, pars = list(boxwex = 0.5, staplewex = 0.25))
```
# t.test의 해석
[http://r-graph.com:3838/ttest](http://r-graph.com:3838/ttest)
# 단정한 데이터(tidy data)
"Happy families are all alike; every unhappy family is unhappy in its own way." –– Leo Tolstoy
"Tidy datasets are all alike, but every messy dataset is messy in its own way." –– Hadley Wickham
## WHO 결핵 데이터
table 1-table 5는 1999-2000년에 WHO에 보고된 Afghanistan, Brazil, China의 결핵환자수 이다.
```{r, message=FALSE}
library(tidyverse)
table1
table2
table3
table4a # cases
table4b # population
table5
```
# Tidy data - 세 가지 규칙(Three rules)
1. 각 변수는 고유한 열에 위치(Each variable must have its own column)
2. 각 관찰치는 고유한 행에 위치(Each observation must have its own row)
3. 각 수치는 고유한 cell에 위치(Each value must have its own cell)

### Tidy data의 잇점
1.일관성 있는 데이터의 구조
2.변수가 열에 위치하고 있기 때문에 R의 장점인 벡터화된 연산이 가능하다.
=> 데이터 분석에 유용한 구조로 dplyr, ggplot2등 tidyverse 패키지들은 모두 tidy data에서 작동한다.
--
예 1) 인구 만 명당 유병률
```{r}
table1 %>%
mutate(rate = cases/population * 10000)
```
---
예 2) 년도별 환자수 총계
```{r}
table1 %>%
count(year,wt=cases)
```
---
예 3) 년도별 환자수 시각화
```{r,fig.height=5}
ggplot(table1,aes(year,cases,colour=country)) +
geom_point() +
geom_line()
```
## Spreading 과 Gathering
- 실제 접할 수 있는 대부분의 데이터는 tidy data가 아니다. 그 이유는 크게 두 가지인데 첫째, 대부분의 사람들은 tidy data의 개념이 없으며 둘째, 데이터는 종종 분석 이외에 다른 목적에 맞게 구조화되어 있기 때문이다. 어떤 데이터는 입력하기 쉬운 구조로 되어 있다.
- 깔끔한 데이타를 만들기 위한 첫번째 단계는 변수와 관측치를 구별하는 일이다. 두번째는 다음과 같은 흔한 문제를 해결하는 것이다.
1. 한 변수가 여러 열에 분산되어 있는 경우
2. 한 관측치가 여러행에 흩어져 있는 경우
=> spread()와 gather()로 해결할 수 있다.
### Gathering
```{r}
table4a
```
table4a의 1999와 2000은 변수의 이름이 아니고 year 변수의 값이고 각 행은 하나의 관측치가 아니라 두개의 관측치이다.

```{r}
table4a %>%
gather(`1999`,`2000`,key="year",value="cases")
```
```{r}
table4b
table4b %>%
gather(`1999`, `2000`, key = "year", value = "population")
```
```{r}
tidy4a <- table4a %>%
gather(`1999`, `2000`, key = "year", value = "cases")
tidy4b <- table4b %>%
gather(`1999`, `2000`, key = "year", value = "population")
left_join(tidy4a, tidy4b)
```
### Spreading
```{r}
table2
```
table2에는 하나의 관측치가 두개의 행에 나누어져 있다. 하나의 관측치는 한 나라, 한 해의 데이터인데 각 관측치가 두 행에 나뉘어져 있다. 이 경우 spread()함수로 데이터를 깔끔한 데이터로 만들 수 있다.
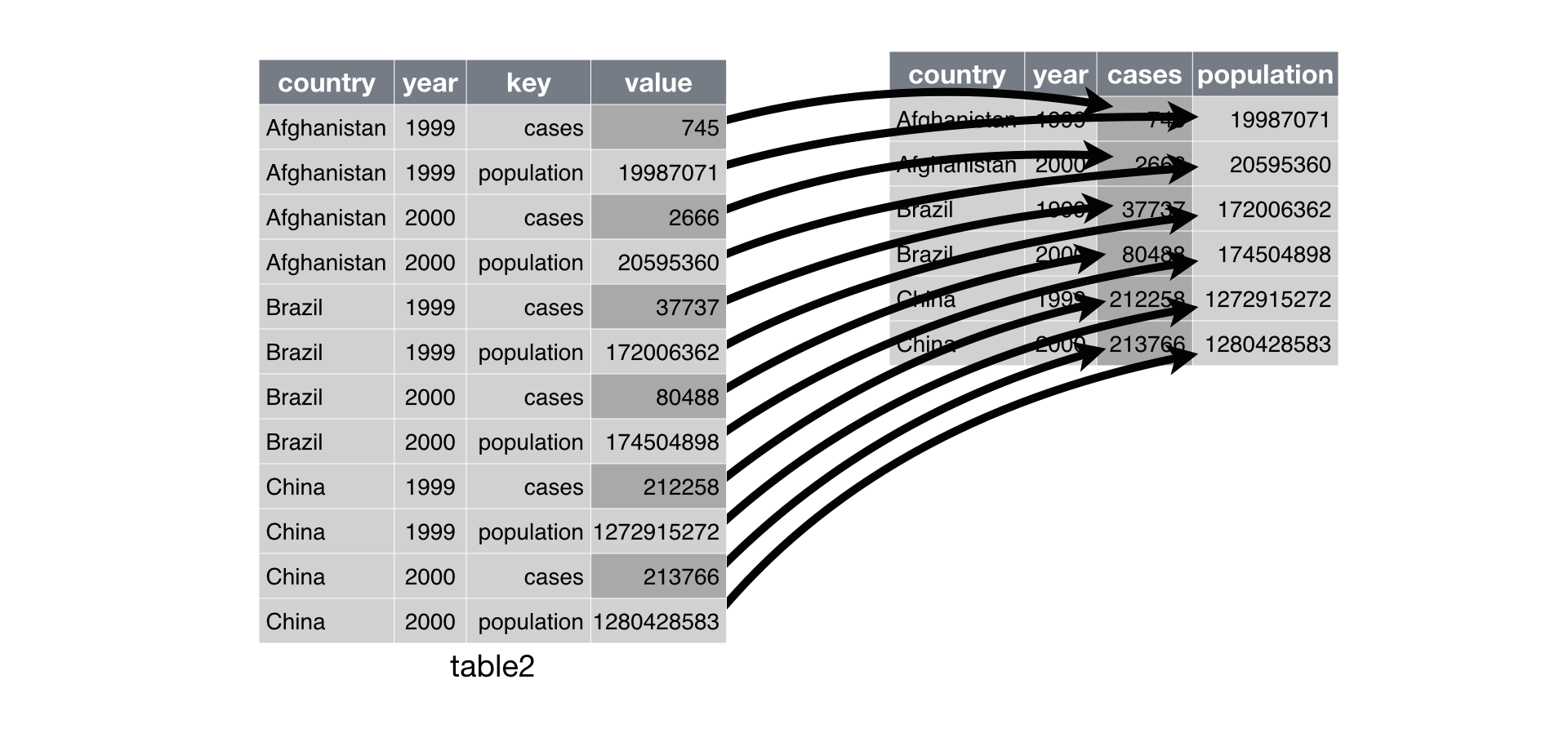
```{r}
table2 %>% spread(key=type,value=count)
```
# long form <-> wide form
학생 10명이 있다. 수업전 test 결과를 pre, 수업후 test결과를 post라고 하자
```{r}
id=1:10
pre=c(70,72,80,85,90,90,60,55,80,90)
post=c(80,73,84,90,95,85,60,50,84,100)
data=data.frame(id,pre,post)
data
```
이 데이타는 전형적인 wide form이다. 이 데이타를 long form으로 바꾸려면 gather()를 사용한다.
```{r}
require(tidyverse)
longdata=gather(data,key=key,value=value,-id)
longdata
```
long form의 데이타를 wide form의 데이타로 바꾸려면 spread()를 사용한다.
```{r}
widedata=spread(longdata,key,value)
widedata
```
# 두 집단의 평균의 비교
1) 정규성검정
- 반응변수가 정규분포 하는 경우 : 모수통계
- 그외 : 비모수통계
정규분포 하는경우
2) 분산이 같은지 검정( var.test() )
3) 분산이 다르면 Welch의 t-test 검정(t.test())
4) 분산이 같으면 pooled variance를 이용한 t-test (var.equal=TRUE)
### 반응변수의 정규성 검정 :
선형모형에 적합시킨후 잔차가 정규분포하는지 검정
```{r}
model=lm(age~sex,data=acs)
shapiro.test(resid(model))
```
```{r}
result=aggregate(age~sex,data=acs,function(x) { c(mean(x),sd(x))})
colnames(result$age)=c("mean","sd")
result
boxplot(age~sex,data=acs)
# 분산이 같은지 F 검정
var.test(age~sex,data=acs)
require(webr)
plot(var.test(age~sex,data=acs))
# 분산이 같으므로 var.equal=TRUE로 검정
result=t.test(age~sex,data=acs,var.equal=TRUE)
result
plot(result)
# 분산이 다르다면 var.equal=FALSE로 검정
t.test(age~sex,data=acs,var.equal=FALSE)
# 비모수검정
wilcox.test(age~sex,data=acs)
```
# 일원분산분석 (One-way ANOVA)
```{r}
aggregate(mpg~cyl,data=mtcars,mean)
boxplot(mpg~cyl,data=mtcars)
# cyl가 numeric이므로 factor변수로 바꾸어준다
mtcars$group=factor(mtcars$cyl)
out=lm(mpg~group,data=mtcars)
out
anova(out)
par(mfrow=c(2,2))
plot(out)
par(mfrow=c(1,1))
```
# 다중비교
```{r}
#install.packages("multcomp")
library(multcomp)
tukey=glht(out,linfct=mcp(group="Tukey"))
summary(tukey)
plot(tukey)
```
# 비모수검정 : Kruskal-Wallis rank sum test
```{r}
kruskal.test(mpg~group,data=mtcars)
```
# 이원분산분석(Two-way ANOVA)
```{r}
boxplot(mpg~group+am,data=mtcars)
boxplot(mpg~am+group,data=mtcars)
with(mtcars,interaction.plot(cyl,am,mpg))
with(mtcars,interaction.plot(am,cyl,mpg))
out=lm(mpg~group+am,data=mtcars)
summary(out)
```
# 공분산분석 (ANCOVA)
```{r}
plot(mpg~wt,data=mtcars,col=cyl)
legend("topright",legend=c("4","6","8"),pch=1,col=mtcars$cyl)
out=lm(mpg~wt+group,data=mtcars)
anova(out)
summary(out)
dunnett=glht(out,linfct=mcp(group="Dunnett"))
summary(dunnett)
library(lattice)
xyplot(mpg~wt|cyl,data=mtcars)
xyplot(mpg~wt|am,data=mtcars)
```
# moonBook package 이용
그룹간의 비교를 한번에 : moonBook 패키지의 이용
```{r}
library(moonBook)
library(ztable)
str(acs)
```
# mytable
```{r}
mytable(sex~age+DM,data=acs)
mytable(sex~.,data=acs)
mytable(sex~.-smoking,data=acs)
res=mytable(Dx~.,data=acs)
res
```
# group변수를 중첩하여 사용
```{r}
mytable(Dx+sex~.,data=acs)
```
# ztable과의 연동
```{r,results='asis'}
z=ztable(res)
print(z,type="html")
```